开源信息:GitHub - AbSciBio/igdesign
许可证:MIT License
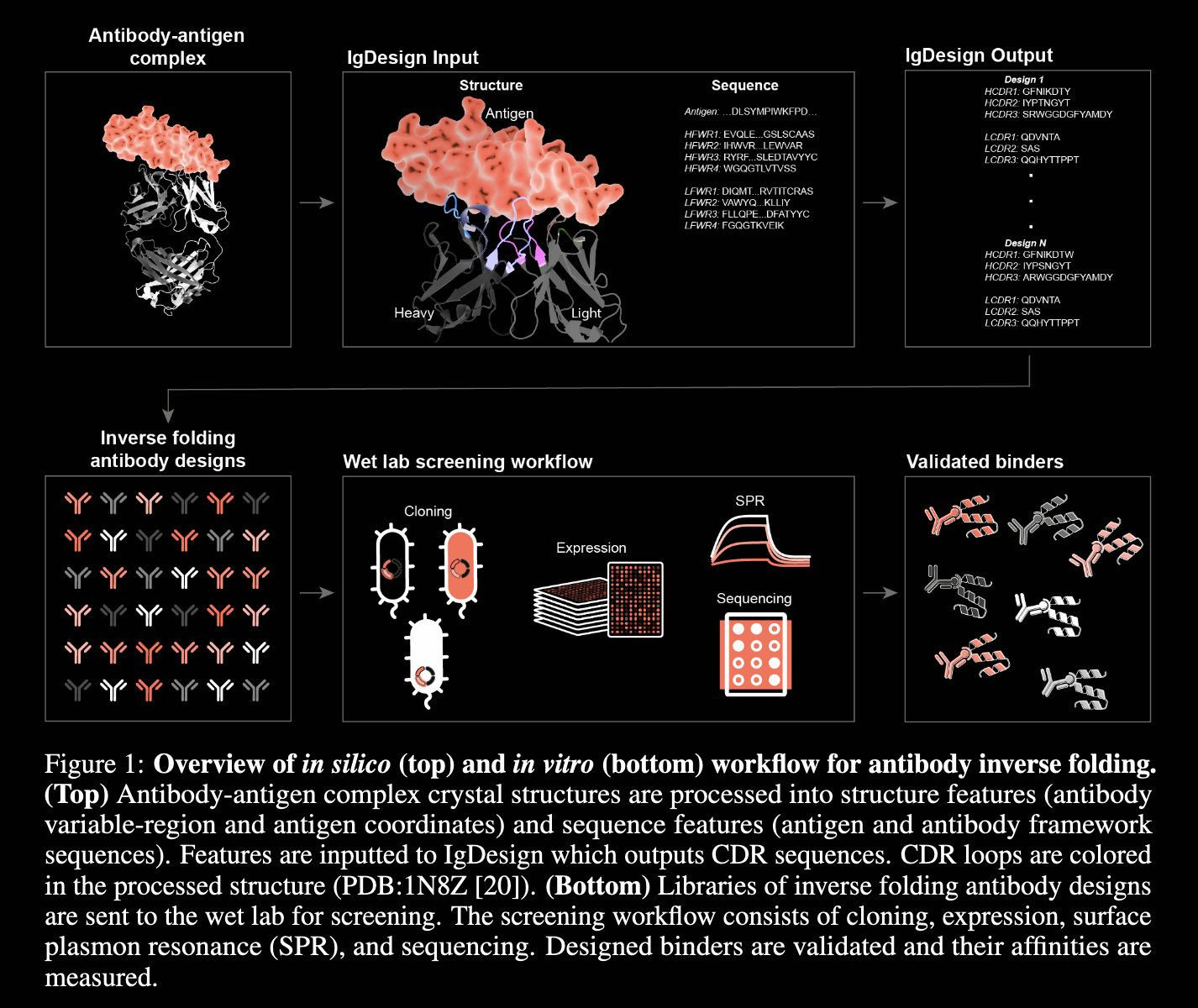
算法解析
Our study introduces IgDesign, a generative antibody inverse folding model based on LM-Design, and the first such model to be validated in vitro for antibody binder design.
IgDesign 是一个基于 LM-Design 的生成抗体反折叠模型,
并首次在体外验证了抗体结合剂的设计。LM-Design是此前字节跳动开源的一个技术路线。
相比于lmdesign使用proteinMPNN原版模型,IgDesign自己训练了一个IgMPNN抗体模型。与类似的抗体模型abMPNN相比,有下列不同:
however we note two key differences between the models: (1) IgMPNN is provided antigen sequence and antibody framework (FWR) sequences as context during training. (2) IgMPNN decodes antibody CDRs in sequential order during training: HCDR1, HCDR2, HCDR3, LCDR1, LCDR2, LCDR3. During inference, any order of CDRs can be specified.
LM-Design的核心思路是利用一个BottleNeck适配器结合结构编码器和PLM解码器,生成更具有天然性质的合理序列。模型选择使用ESM2-3B作为PLM模型,并自行训练proteinMPNN抗体版本IgMPNN,最终干湿结合筛选得到阳性序列。
IgMPNN信息比较如下:
| 参数 | IgMPNN | proteinMPNN |
|---|---|---|
| k_neighbors | 48 | 64 |
| features | (1) Cα-Cα原子之间的距离,(2) 相对Cα-Cα-Cα框架的方向和旋转,(3) 主链二面角,(4) 确定相对链位置的二元特征,(5) 相对位置编码 | 使用16个径向基函数(RBF)对i和j残基之间的N、Cα、C、O以及虚拟Cβ的距离进行编码,这些RBF在2Å到22Å之间等距分布。 |
Our featurization differs from [1] in two ways: (1) We do not assume access to any side chain atoms and thus we do not include any pairwise distance features involving side chain atoms. (2) We include embedded residue type features for all antigen residues and antibody framework residues. We replace the antibody CDR residue embeddings with zero vectors.
如其原文所述,IgMPNN的特征化与proteinMPNN不同,IgMPNN的特征不包括侧链原子,但额外编码了残基类型。ProteinMPNN认为节点特征没有必要而没有加入,但IgMPNN通过引入残基类型或许更有利于局部设计。
训练数据
数据来源
- RCSB PDB数据:预训练IgMPNN模型
- SAbDab复合物数据 (2022.12.06):训练IgMPNN模型和组合构建的IgDesign模型
数据划分
- 训练集划分
为避免数据泄露,PDB依据序列相似度40%聚类划分,Sabdab依据抗原序列相似度40%划分。
- 参考分子选择
超参数
- 优化器:Adam
- 学习率:1e-3
实验验证
库筛
从sabdab中选择8个抗原,依次使用IgDesign设计HCDR3,HCDR1,HCDR2,LCDR1,LCDR2,LCDR3。
每个抗原生成一百万序列,然后筛选出CE损失最小的100条HCR3设计序列和HCDR123设计序列进行湿实验。
湿实验
从E. coli中表达抗体,SPR实验检测抗体与抗原的结合力,并测序确定抗体序列。
结果
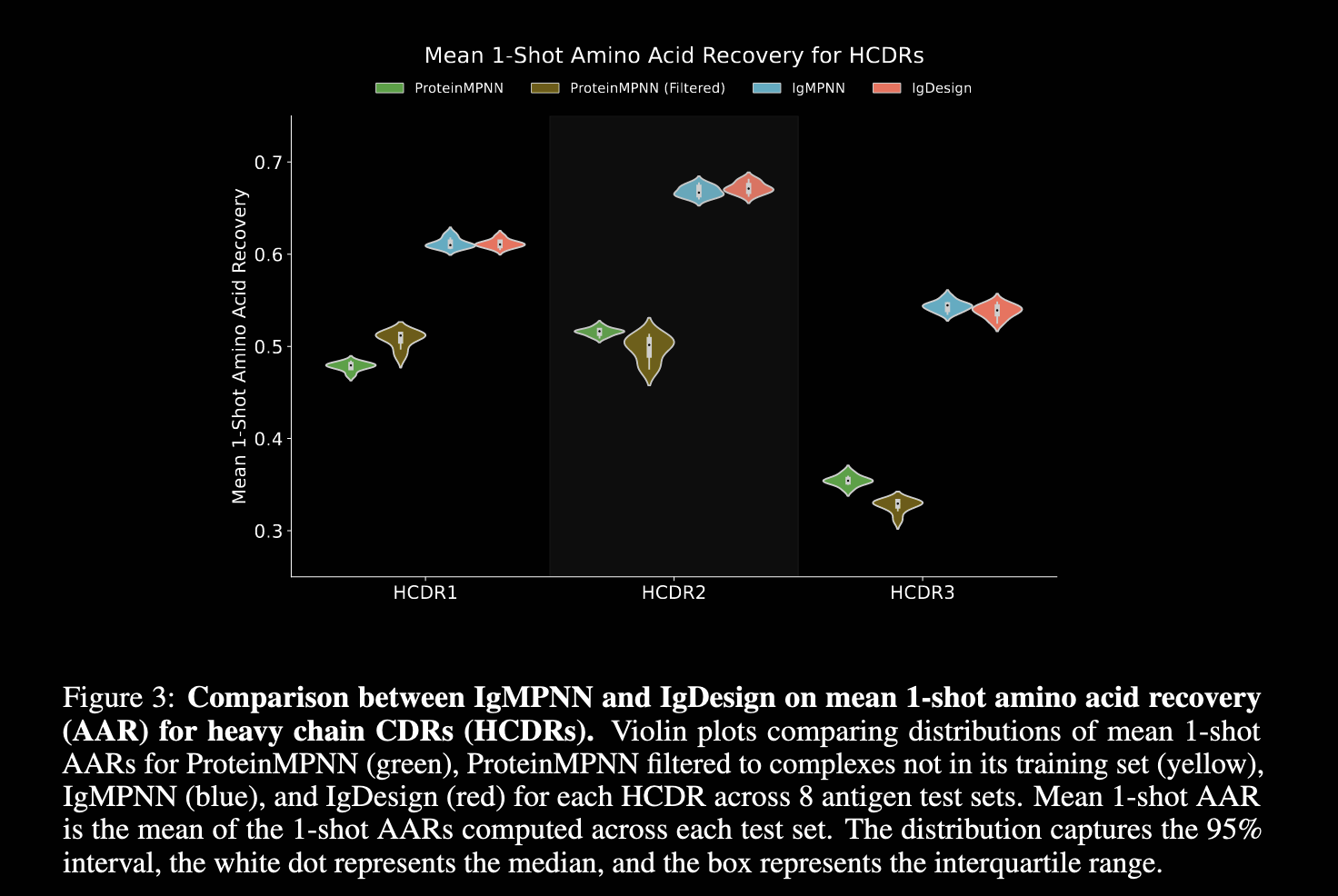
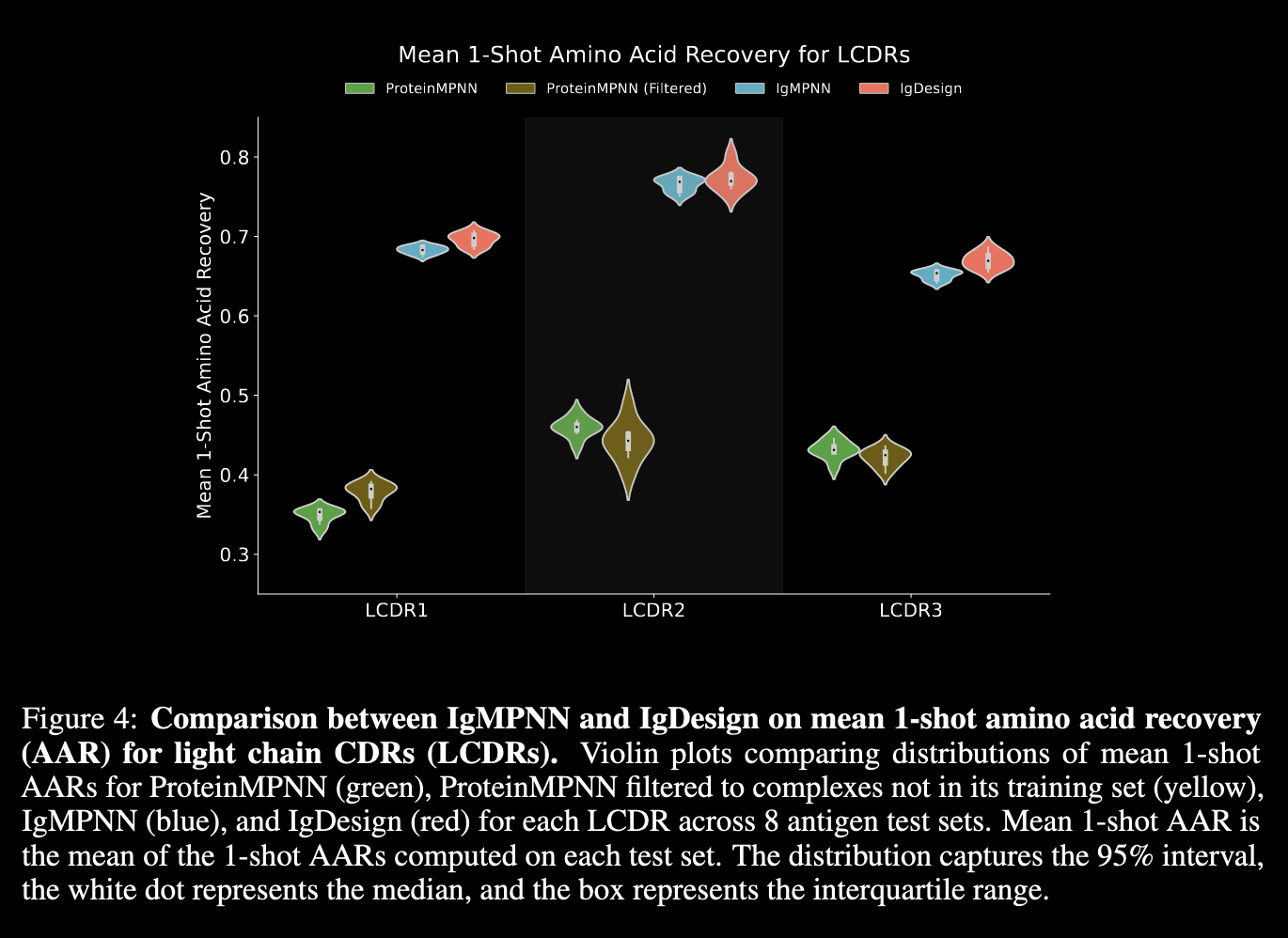
- AAR
下图是对比proteinMPNN、IgMPNN、IgDesign的AAR结果。
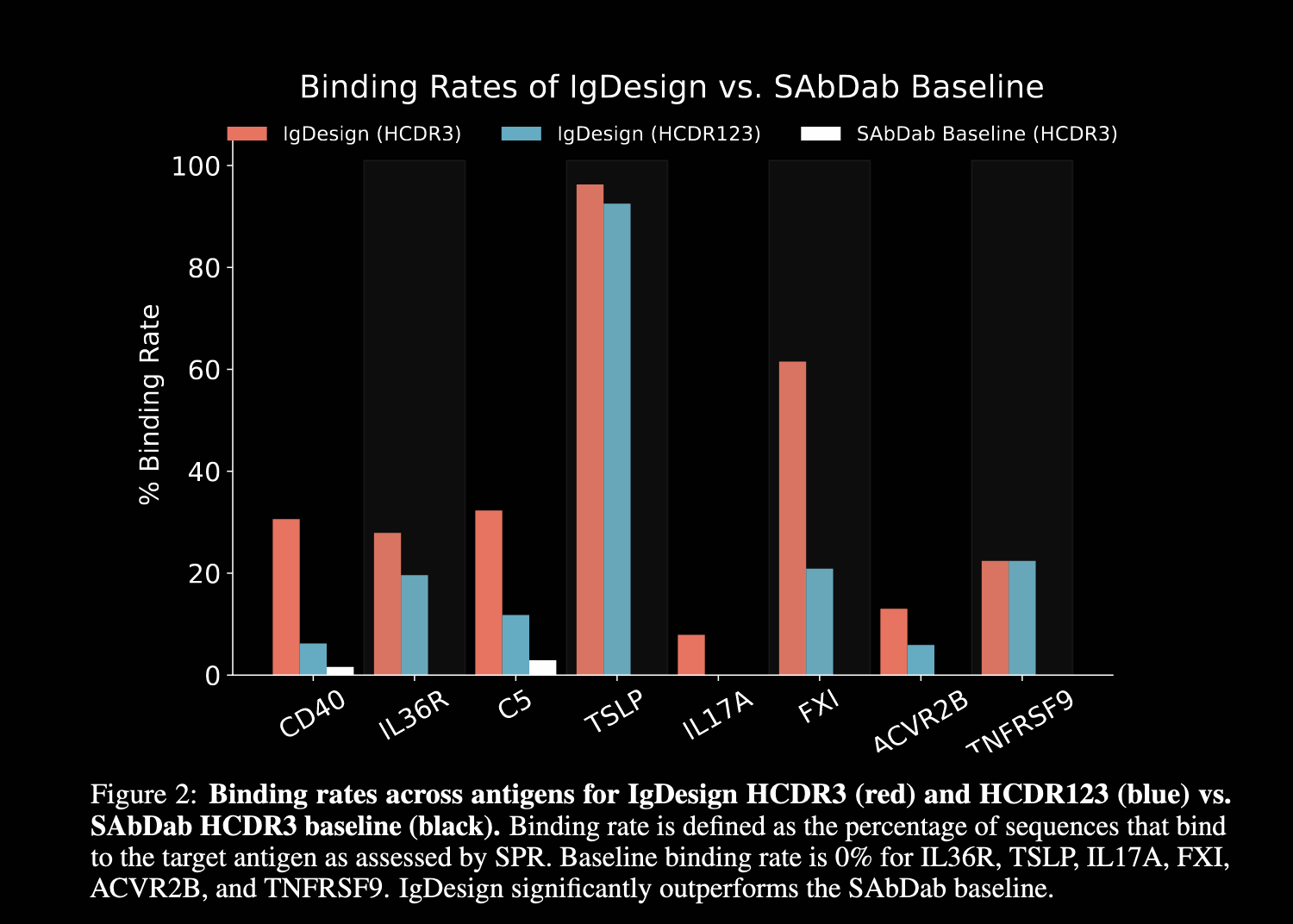
- SPR结合率
相比从sabdab随机采样,IgDesign设计抗体结合率更高。

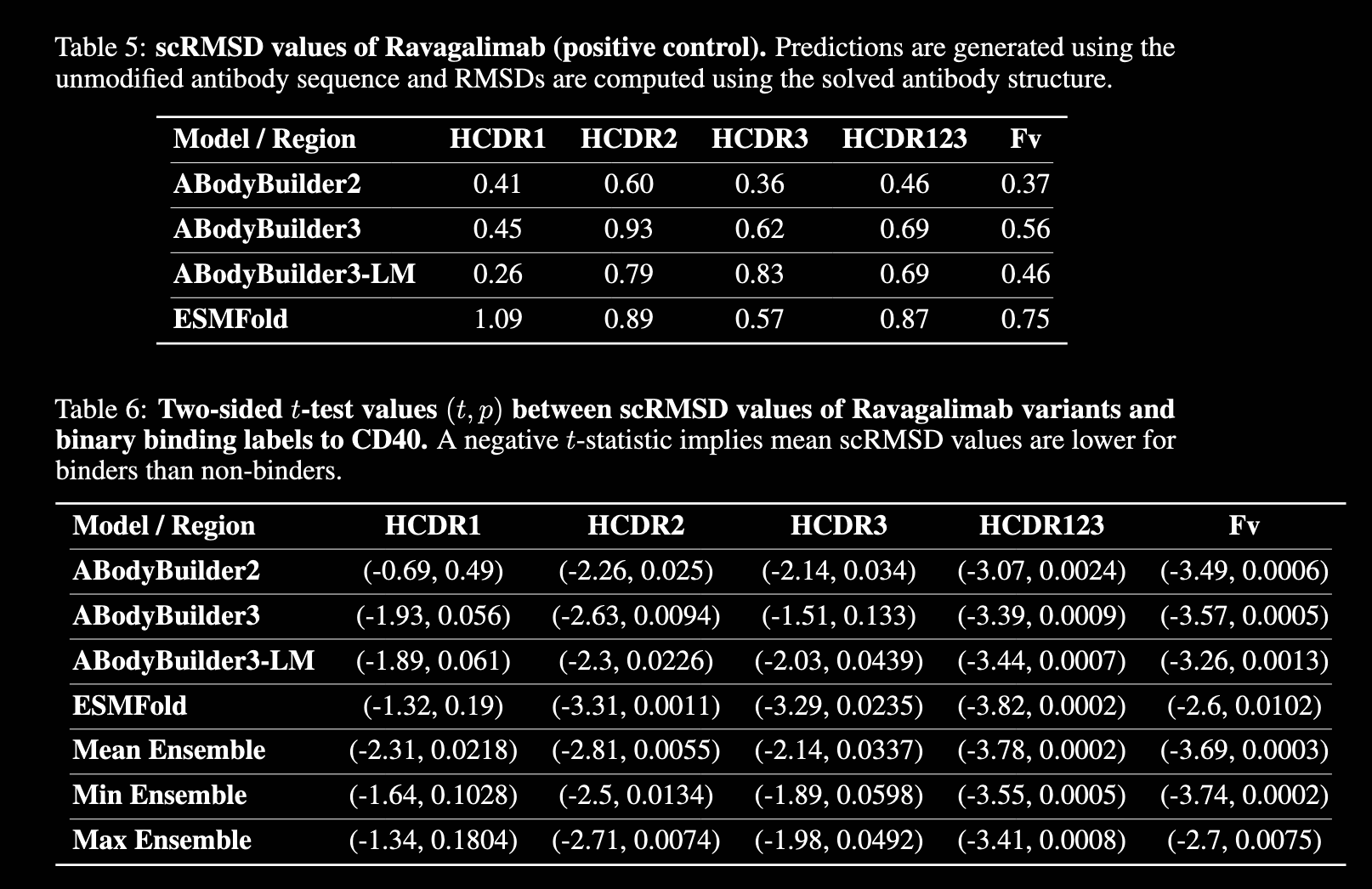
- scRMSD
本文用了多个不同结构预测模型来评估scRMSD,并使用双端t-test来评估显著性。
与wt分布做比照,binding的scRMSD分布t值相对低于non-binding。下面是Ravagalimab/CD40的结果。
笔者看法
IgDesign的方案实际上是对LMDesign应用于抗体设计的一个尝试,单从给出的SPR结果来看,
效果挺不错,部分靶点结合率很高。
但AbSci对环节中最至关重要的序列筛选环节没有给出更多的信息,
只依据CE损失函数筛选的可信度较低,
一般来说这个筛选需要结合多种计算和专家经验。
此外,IgDesign的比较对象较为单调,没有和AbMPNN、LMdesign等相关模型做横向对比,
反而选择了基于sabdab采样等baseline比较,是没有什么意义的。