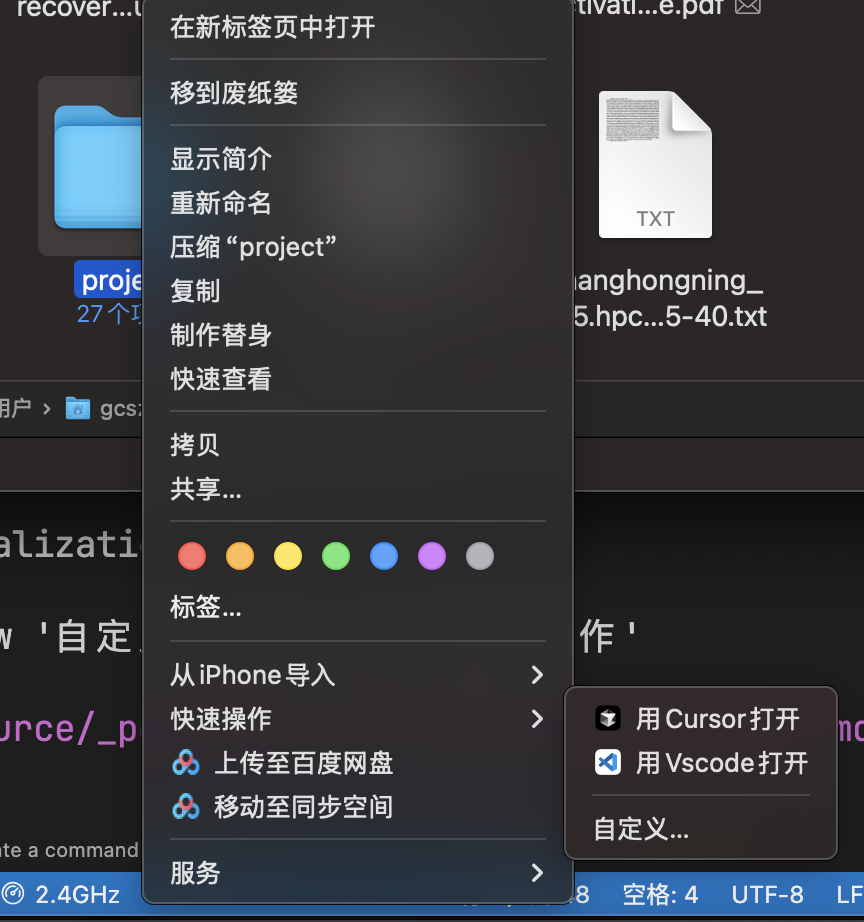
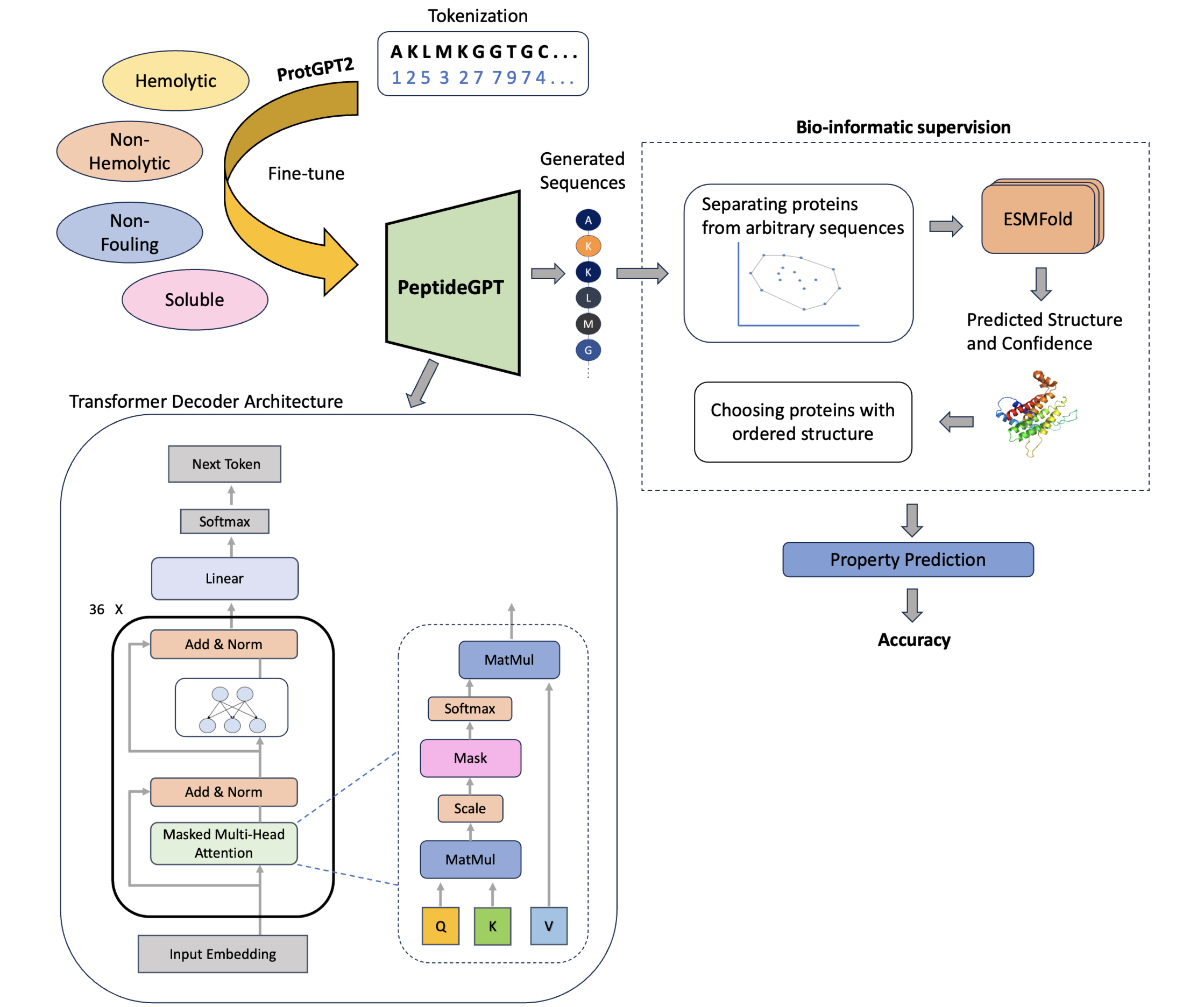
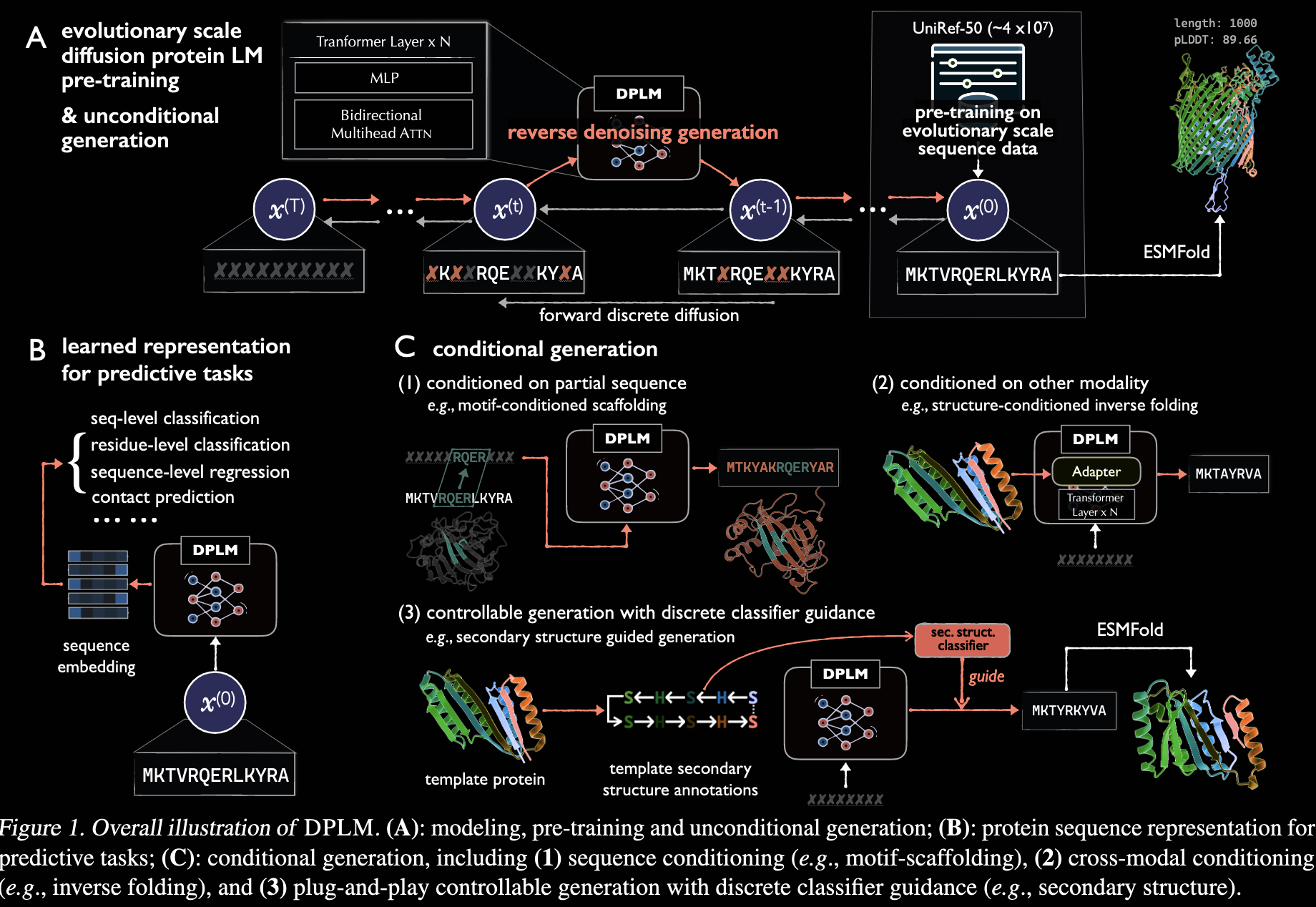
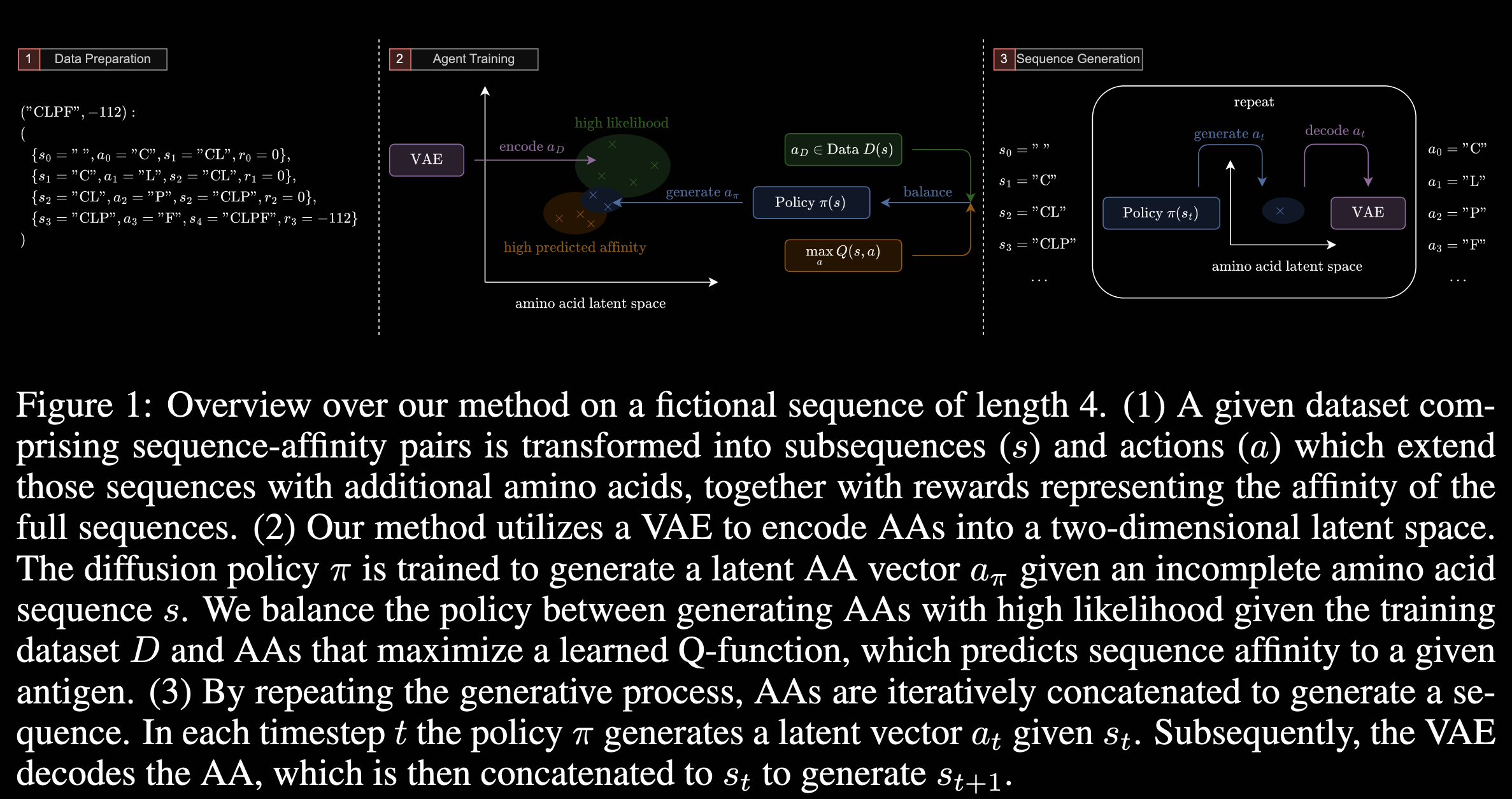
右键菜单的配置可以通过修改注册表实现。其基本项架构是如下配置,其中 command 参数可以是 %1 或 %V 或 %V %1 或 %V %1 %2 等。不同位置右键的配置位置不同,可以分为三类。
1
2
3
4
5
| XXX/
(Default)='通过 XXX打开'
Icon='C:\Users\Administrator\Desktop\XXX.exe'
command/
(Default)="`"$exe_path`" `"%1`""
|
选中文件时的右键菜单:
相关内容在 Registry::HKEY_CLASSES_ROOT\*\shell 下。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
| $exe_path='C:\Users\Administrator\Desktop\Cursor.exe'
$program_name='Cursor'
$message='通过Cursor打开'
$reg_base_path='Registry::HKEY_CLASSES_ROOT\*\shell'
$reg_path="$reg_base_path\$program_name"
$reg_command_path="$reg_path\command"
if (-not (Test-Path $reg_path)) {
New-Item $reg_path -Force
}
Set-ItemProperty -Path $reg_path -Name '(Default)' -Value $message
New-ItemProperty -Path $reg_path -Name 'Icon' -Value $exe_path
New-Item -Path $reg_command_path -Force
Set-ItemProperty -Path $reg_command_path -Name '(Default)' -Value "`"$exe_path`" `"%1`""
|
选中文件夹时的右键菜单:
相关内容在 Registry::HKEY_CLASSES_ROOT\Directory\shell 下。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
| $reg_base_path='Registry::HKEY_CLASSES_ROOT\Directory\shell'
$program_name='Cursor'
$message='通过Cursor打开'
$reg_path="$reg_base_path\$program_name"
$reg_command_path="$reg_path\command"
if (-not (Test-Path $reg_path)) {
New-Item $reg_path -Force
}
Set-ItemProperty -Path $reg_path -Name '(Default)' -Value $message
New-ItemProperty -Path $reg_path -Name 'Icon' -Value $exe_path
New-Item -Path $reg_command_path -Force
Set-ItemProperty -Path $reg_command_path -Name '(Default)' -Value "`"$exe_path`" `"%1`""
|
选中文件夹背景时的右键菜单:
相关内容在 Registry::HKEY_CLASSES_ROOT\Directory\Background\shell 下。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
| $reg_base_path='Registry::HKEY_CLASSES_ROOT\Directory\Background\shell'
$program_name='Cursor'
$message='通过Cursor打开'
$reg_path="$reg_base_path\$program_name"
$reg_command_path="$reg_path\command"
if (-not (Test-Path $reg_path)) {
New-Item $reg_path -Force
}
Set-ItemProperty -Path $reg_path -Name '(Default)' -Value $message
New-ItemProperty -Path $reg_path -Name 'Icon' -Value $exe_path
New-Item -Path $reg_command_path -Force
Set-ItemProperty -Path $reg_command_path -Name '(Default)' -Value "`"$exe_path`" `"%V`""
|