PPB-Affinity:整合多个数据源的PPI亲和力数据集
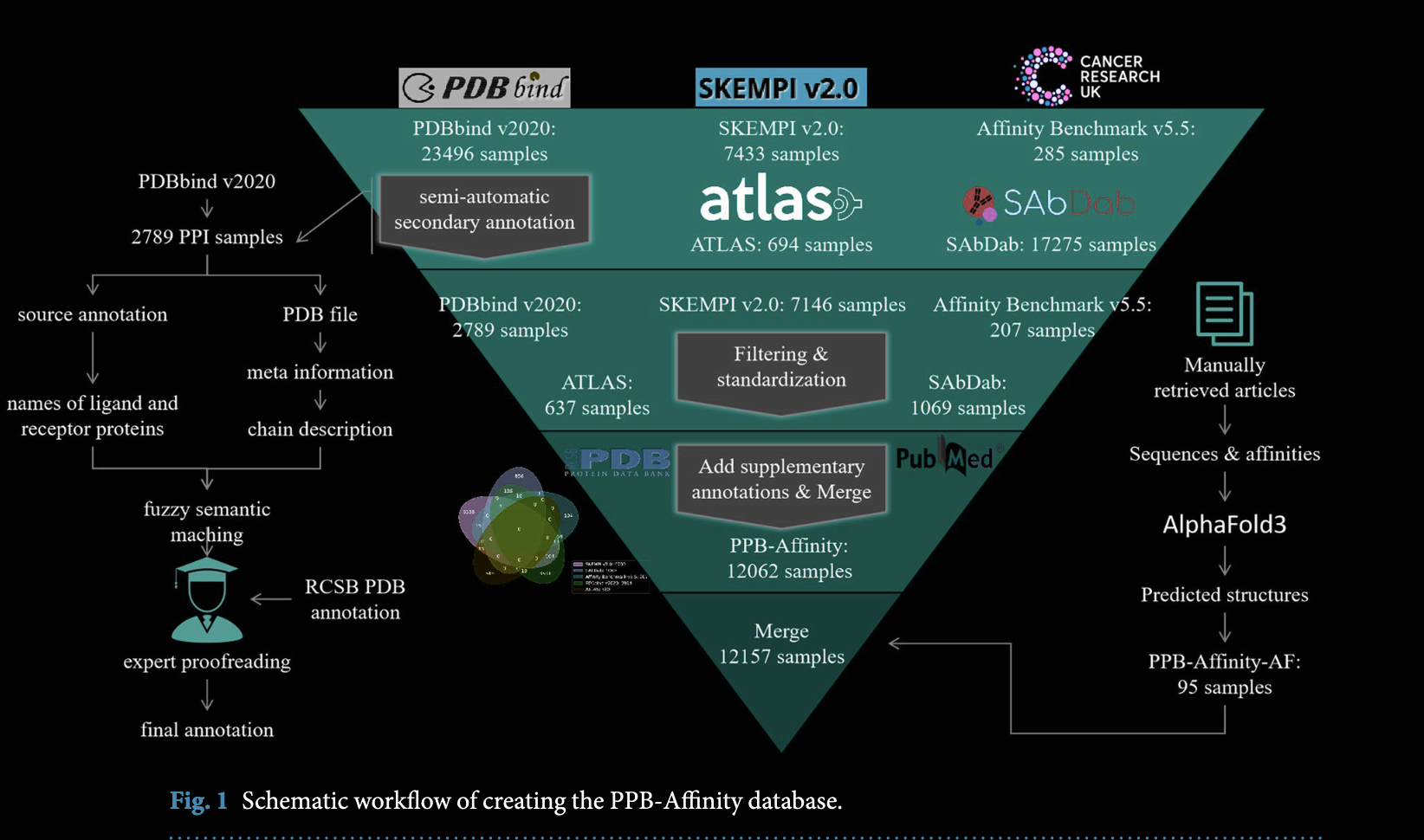
2024年12月3日,清华大学珠三角研究院人工智能创新中心的韩蓝青研究团队在Nature Scientific Data上在线发表了PPB-Affinity: Protein-Protein Binding Affinity dataset for AI-based protein drug discovery,整合了SKEMPI v2.0、PDBbind 2020和SabDab等数据源,整理构建一个PPI亲和力数据集,并基于IPA构建一套benchmark算法验证了数据集的价值。
一、开源信息
- 数据集pipeline:https://github.com/Huatsing-Lau/PPB-Affinity-DataPrepWorkflow
- benchmark算法:https://github.com/ChenPy00/PPB-Affinity
- 数据地址:https://zenodo.org/records/14271435
- 模型地址:https://huggingface.co/ChenPy00/PPB-Affinity
二、数据统计概括
- 12062条晶体结构数据,3032个unique PDB code。
- 数据分布:
- SKEMPI v2.0:6700 (55.55%)
- PDBbind v2020:3600 (29.85%)
- SAbDab:1055 (8.75%)
- ATLAS:545 (4.52%)
- Affinity Benchmark v5.5:162 (1.34%)
- 检测方法:
- SPR:3618 (30.00%)
- FL:1458 (12.10%)
- ELISA:172(1.43%)
- BLI:104 (0.86%)
- ITC:591(4.90%)
- SP:336 (2.79%)
- RA:320(2.65%)
- IARA:125(1.04%)
- IAGE:9(0.07%)
- Other:805(6.67%)
- Unknown:254(2.11%)
- N/A:3765(31.21%)
- affinity的值($pKD = -\log_{10}(KD)$):
- total:7.54±2.14,max=15.70,min=1.32
- SPR:6.92±2.05,max=13.80,min=2.29
- 晶体解析方法
- X-RAY DIFFRACTION:11722 (97.18%)
- SOLUTION NMR:300 (2.48%)
- ELECTRON MICROSCOPY:30 (0.24%)
- 分辨率(Å):2.40±0.65
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 潇洒记忆!