DPLM扩散蛋白语言模型-字节跳动ICML2024
原文链接:Diffusion Language Models Are Versatile Protein Learners
开源信息:GitHub - bytedance/dplm,Apache 2.0 License。
DPLM是字节跳动在ICML2024上提出的一种基于扩散的语言模型,用于蛋白质序列的生成。
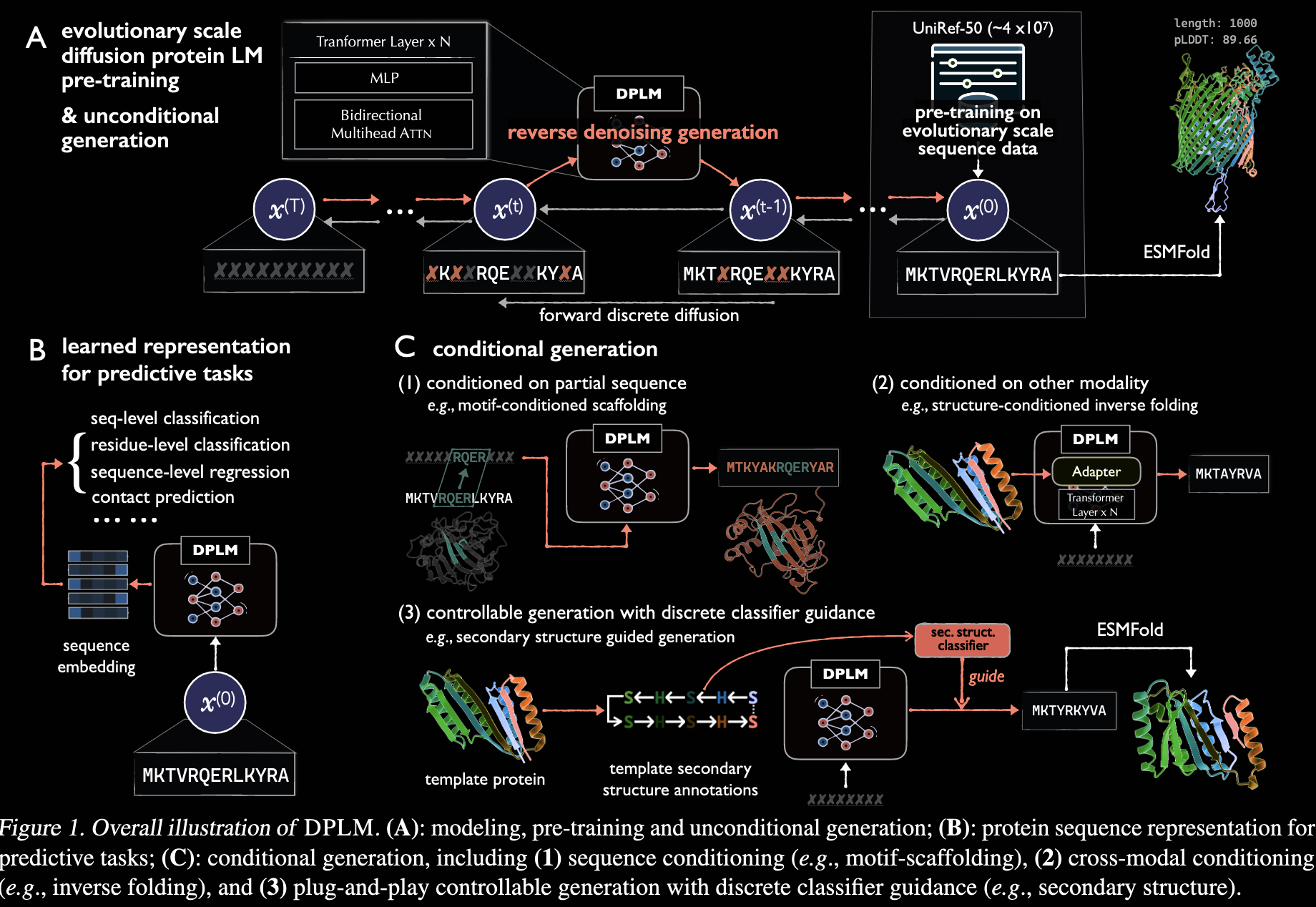
算法解析
蛋白序列由20种天然氨基酸组成,因此假设蛋白序列$x$服从分类分布,
即为$x\sim \text{Cat}(x; p)$,其中$p$是蛋白序列的类别分布。
对于离散分布的扩散,其forward过程是一个离散分布转移的马尔可夫过程。
可以用下列公式表示:
反向过程则是利用了PLM进行概率分布的逆向估计,训练过程中会约束相同时间步长的正反向过程的分布一致(KL散度)。
同时,由于从头直接训练PLM+扩散模型存在困难,因此采用了预训练的PLM作为初始化,然后进行扩散模型的训练。而且作者将PLM训练的MASK比例一开始设置较小,逐渐增加来提升模型起初的学习效果。
实验结果
作者在多个下游场景中进行了实验。
无条件序列生成
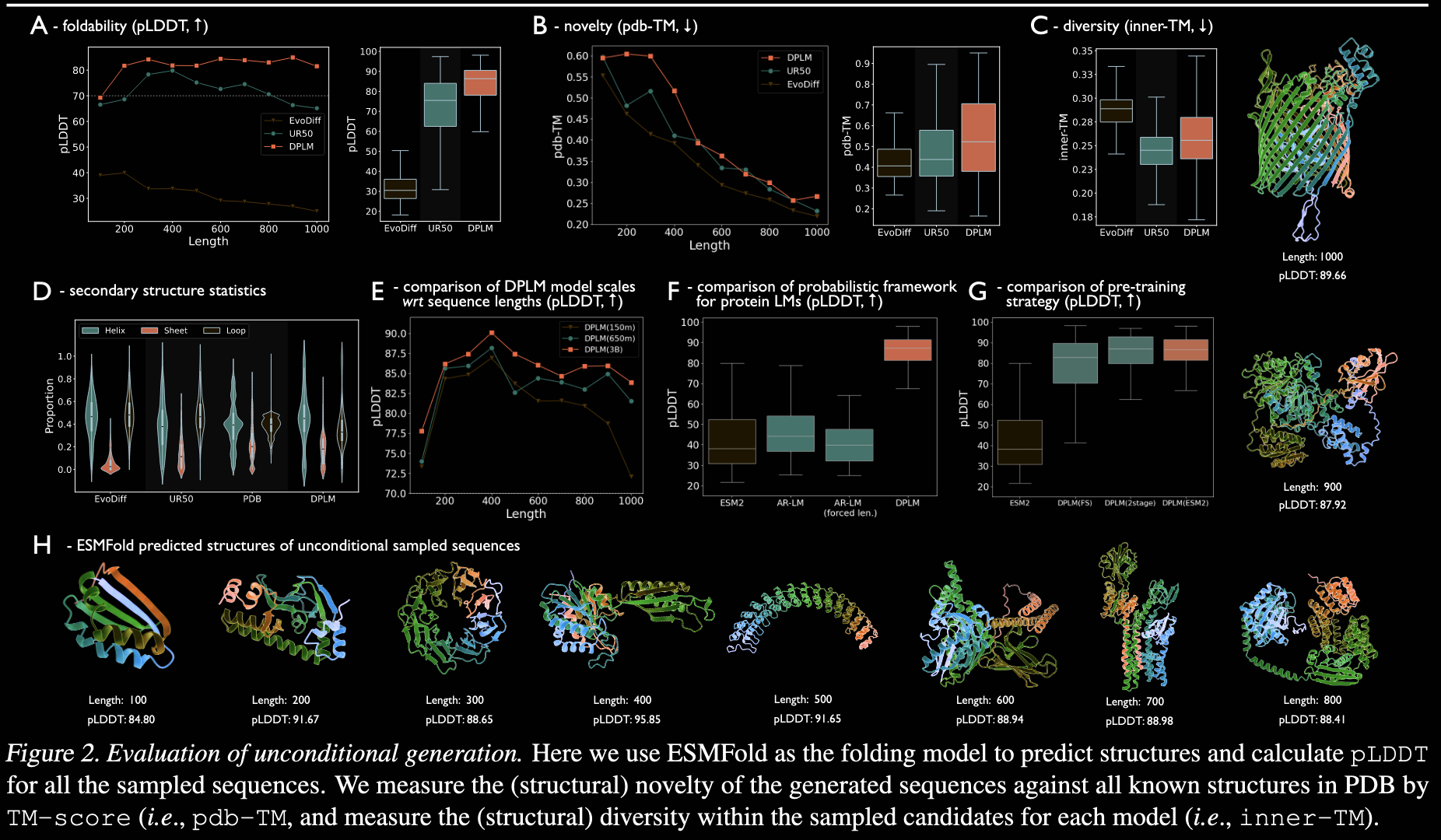
没有任何条件限制下进行了序列设计,然后使用ESMFold进行结构预测,分别评估了下列指标:
- foldability:使用ESMFold结构预测,根据pLDDT值判断性能,算法表现较好。
- novelty:与PDB中结构的TMscore,越小越好,算法表现一般。
- diversity:多样性,设计序列内的相互TMscore,越大越好,算法表现较好。
基于PLM表征层的蛋白功能和性质预测
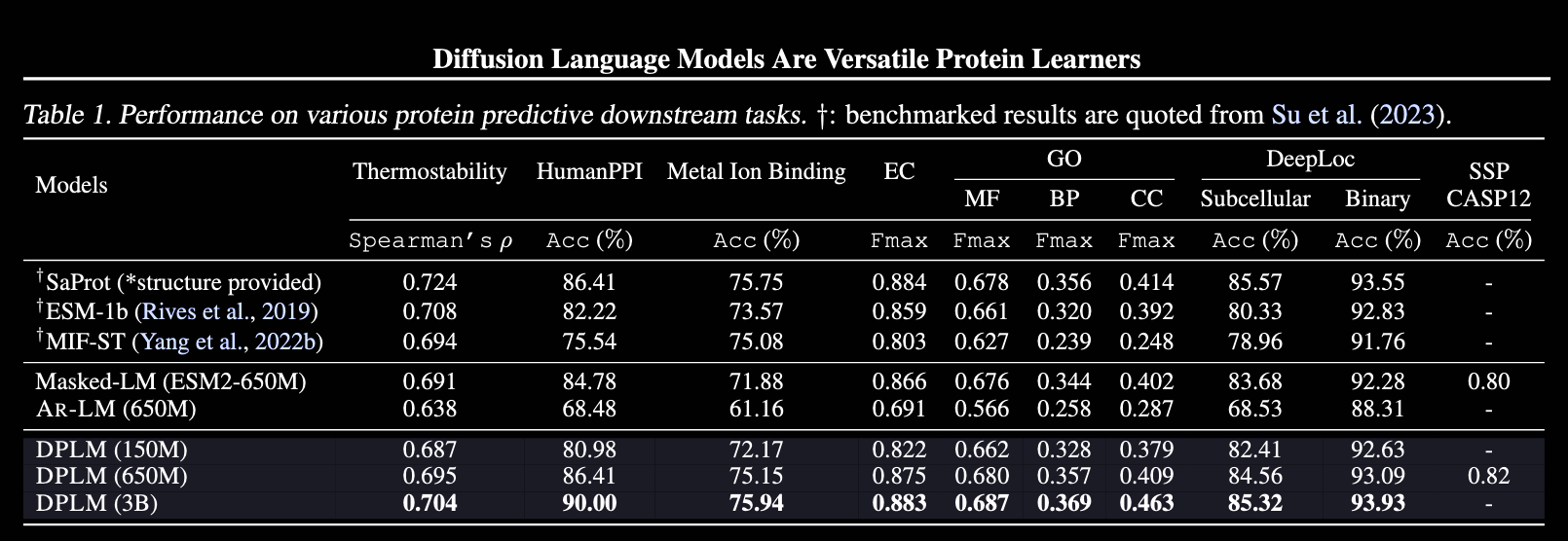
PLM的一个重要应用就是蛋白功能和性质的预测,作者将其应用到热稳定性、PPI、金属离子结合和GO术语分类等任务中,均取得了较好的效果。但是也需要注意他所比较的模型也是属于PLM的范畴,实际上性能不能超过很多专门设计的模型,如GO功能注释的AnnoPRO。
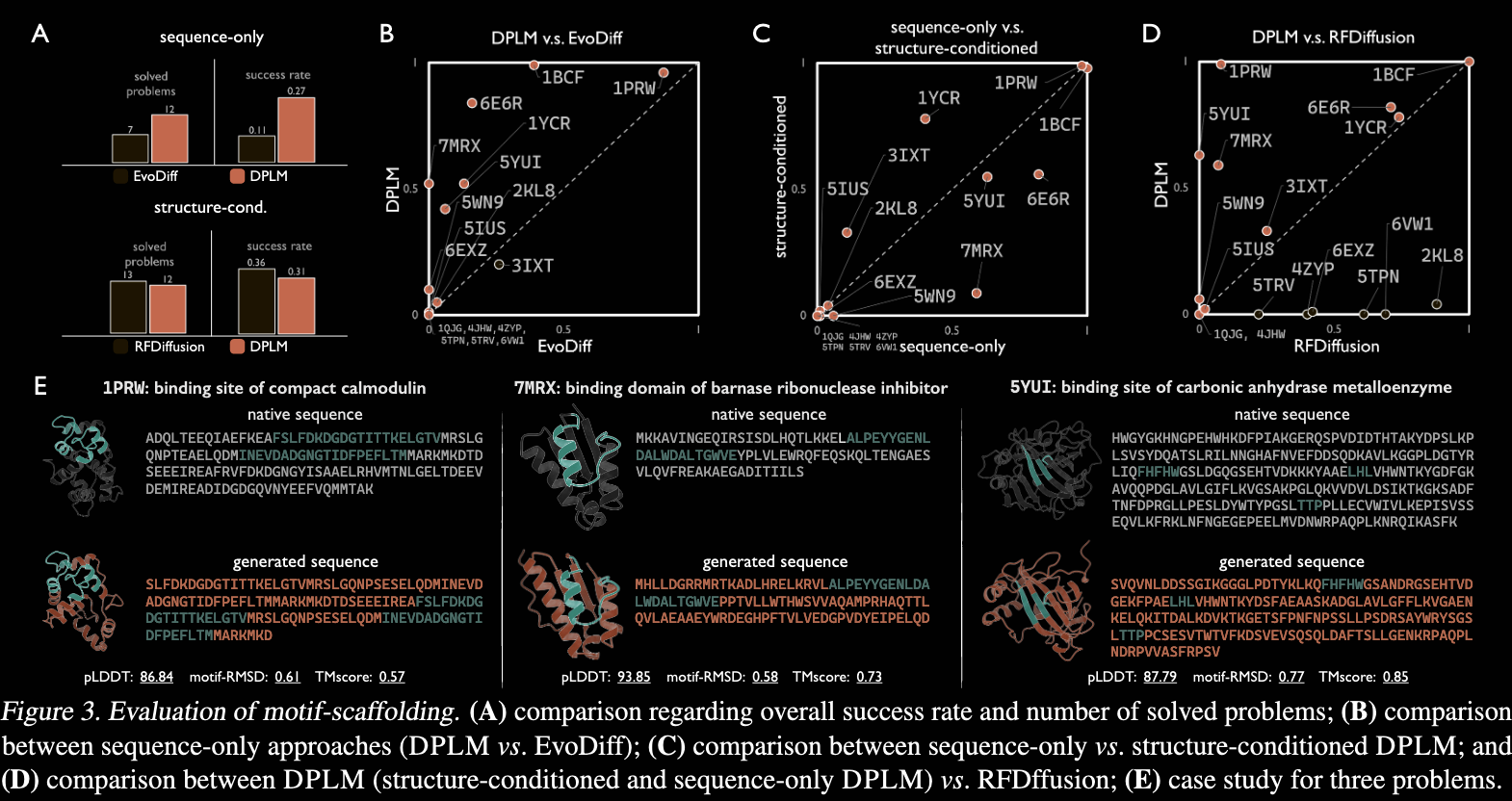
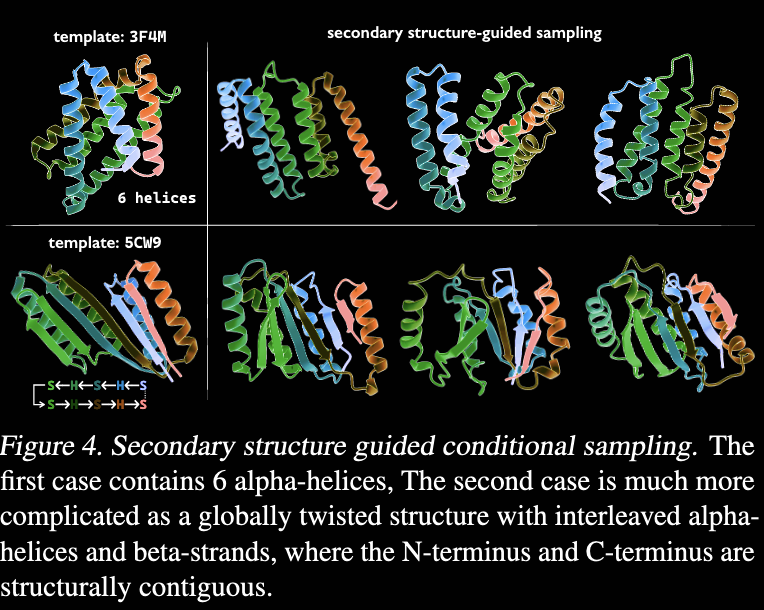
给定条件下的序列生成
这里的条件可以分为两种:
- 给定序列上下文,进行序列生成,比较适用于motif-scaffold设计。
- 给定结构,进行序列设计。
前者是PLM的固有能力,而后者则需要整合结构模块。作者利用了此前的工作ByProt,将结构模块整合到PLM中,然后进行序列生成。
相比于base模型,DPLM总体指标有提升。

本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 潇洒记忆!