论文名称:《Retrieval Augmented Diffusion Model for Structure-informed Antibody Design and Optimization》
论文亮点:利用MASTER算法,通过输入给定CDR骨架结构,检索PDB得到相似的CDR fragment数据,从而增强序列设计的能力。
论文缺点:检索增强的PDB可能搜到原始数据,因此存在数据泄露风险。对未能检索groud truth的数据性能较差。
论文代码:暂无开源信息。
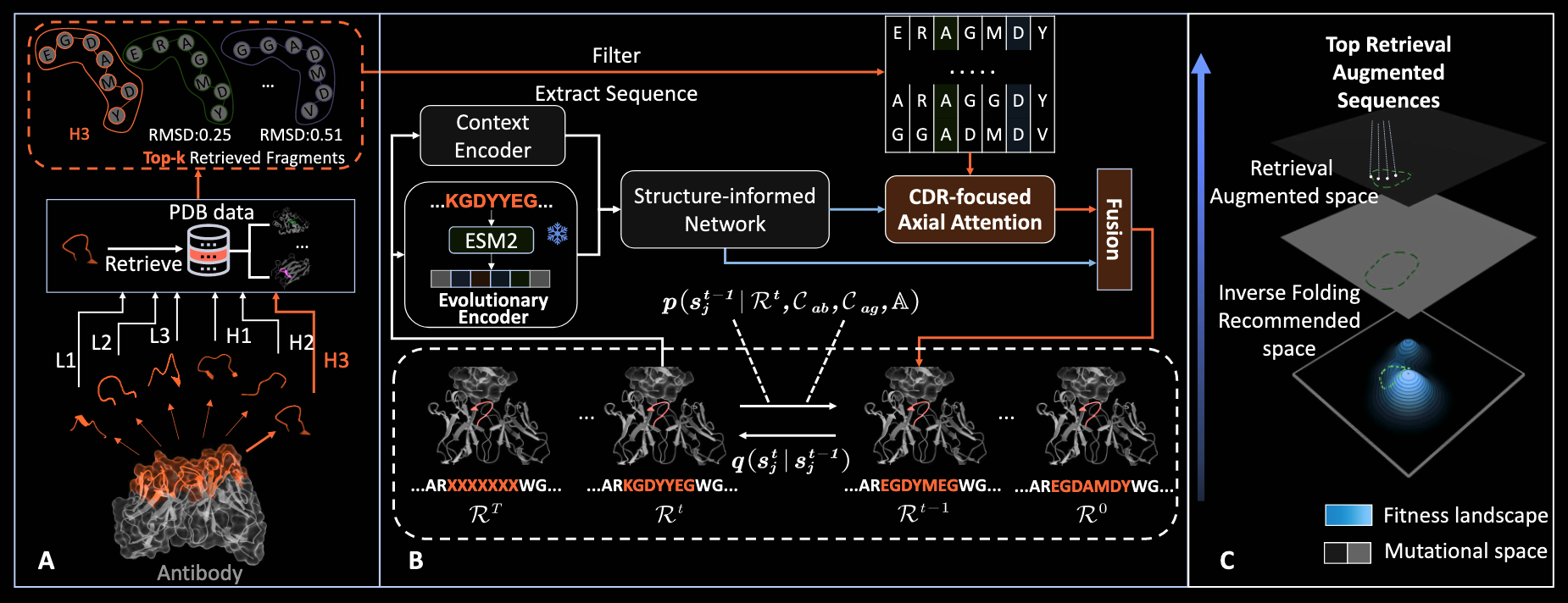
算法解析
RADAb算法是一个基于RA信息的序列扩散算法,这与RFdiffusion这类结构扩散算法不同。
变量声明
在文章开篇,作者对其声明的算法变量进行了说明:
- 第$i$位的残基类型$s_i\in {ACDEFGHIKLMNPQRSTVWY}$
- 第$i$位的残基CA原子坐标$x_i \in R^3$
- 第$i$位的残基原子相对标准位置的旋转矩阵$O_i\in SO(3)$
- $i=1,2,…,N$,其中N为序列长度。
- $R={s_j|j\in {a+1,…,a+m}}$为位于$a+1$和$a+m$区间的需要设计的CDR3序列采样分布。
- $C_{ab}={(s_i,x_j,O_j)|i\in{1,…,M}\backslash{a+1,…,a+m},j\in{1,…,M}}$表示给定抗体结构和非CDR残基序列信息。
- $C_{ag}={(s_i,x_j,O_i)|i\in{M+1,…,N}}$表示给定抗原的结构和序列信息。
- $\mathbb{A}={A_i|i\in{1,…,k}}$表示检索到的CDR fragment信息。
最终目标就是预测CDR序列结合$R$的分布。
扩散模型定义
这里的扩散模型是序列的扩散模型,结构化信息是在模型中嵌入。
前向传播过程,线下求和系数$\beta^t$会随时间逐渐从0增加到1,不断增加随机噪声,直到天然氨基酸的随机分布。
反向传播过程就是模型训练的目标,能够扩散生成序列,而前面的结构信息和当前分布会通过给定的神经网络$F(.)[j]$嵌入反向传播。
这里采用了DiffAb的架构作为一个扩散模型。
检索算法
论文采用MASTER算法进行fragment查询,使用RMSD进行约束。算法描述比较笼统,但简单来说就是沿着残基进行长度为m的扫描,计算RMSD,如果符合要求则算到里面。

虽然MASTER算法不考虑序列同源性,但作者在训练时对搜到的序列片段进行了identity分析,保留了同源性较高的片段用来训练(作为进化信息)。但在推理生成时没有进行筛选。
扩散生成模型架构
如其图所示,$F$模型存在两个分支,一个负责接收$Rt, C{ab}, C_{ag}$三块全局的结构和序列信息的全局几何信息分支,一个负责接收RA检索信息的局部CDR信息分支。
全局几何信息分支
这一分支分为一个结构编码模块、一个序列编码模块以及一个信息整合模块。
- 结构编码:分别有由两个MLP负责生成残基自身特征编码$zi$和残基相互之间几何关系特征编码$y{ij}$。
- 序列编码:就是用当前的序列PSSM矩阵信息输入ESM2-650M的模型,拿到一个embedding信息$e_t$。
- 信息接收模块:上述结构编码和序列编码后送入这个模块,拼接输入一个IPA(invariant point attention)模块转化为隐变量$h_i$,然后通过MLP转换为当前timestamp的一个$R^{t-1}$预测概率分布。
局部CDR信息分支
这里实际上可以把检索的CDR片段信息看作类似alphafold的MSA信息,作者会将第一个分支给出预测分布与MSA信息进行类似MSA Transformer那样的row attention与column attention计算,得到一个CDR-focused axial attention信息矩阵,最后矩阵转换为加入检索信息的PSSM概率矩阵。
同时模型架构加入了残差设计,希望将少因此带来的结构信息损失。
相比与直接进行序列MSA,结构相似性搜索更有利于增加设计CDR的多样性。但是作者又将训练时MSA进行做了相似性筛选,这是比较令人困惑的。
todo:深挖一下里面可能的计算实现细节。
Loss的定义
每一步的loss是计算了每个位置残基概率分布的KL散度
总的loss就是每个步的loss的期望:
下面是官方给的训练伪代码:
训练配置
- 框架:Pytorch
- 优化器:Adam
- 学习率:1e-4
- 学习率优化:employed plateu (reduce rate 0.8)
结果评估
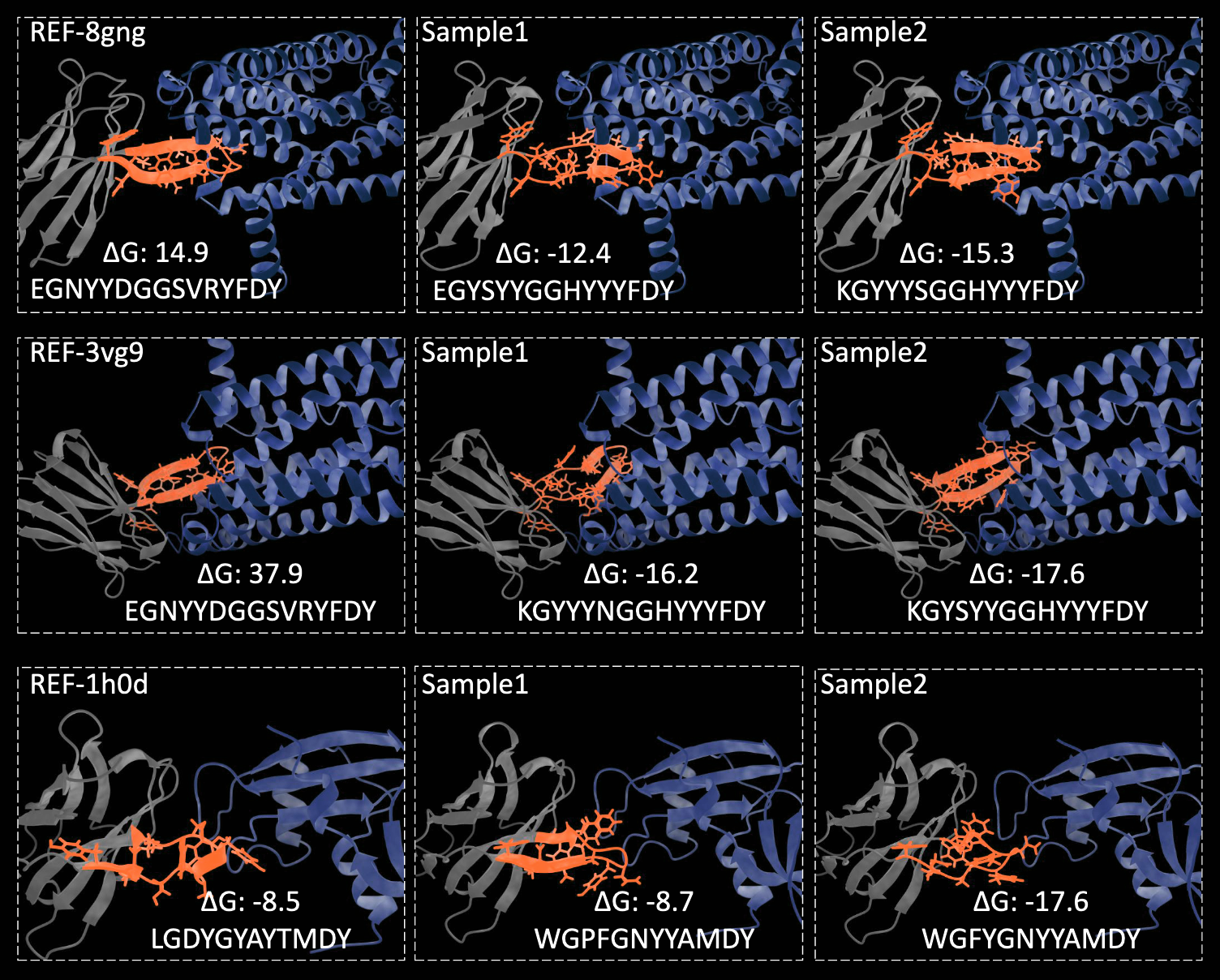
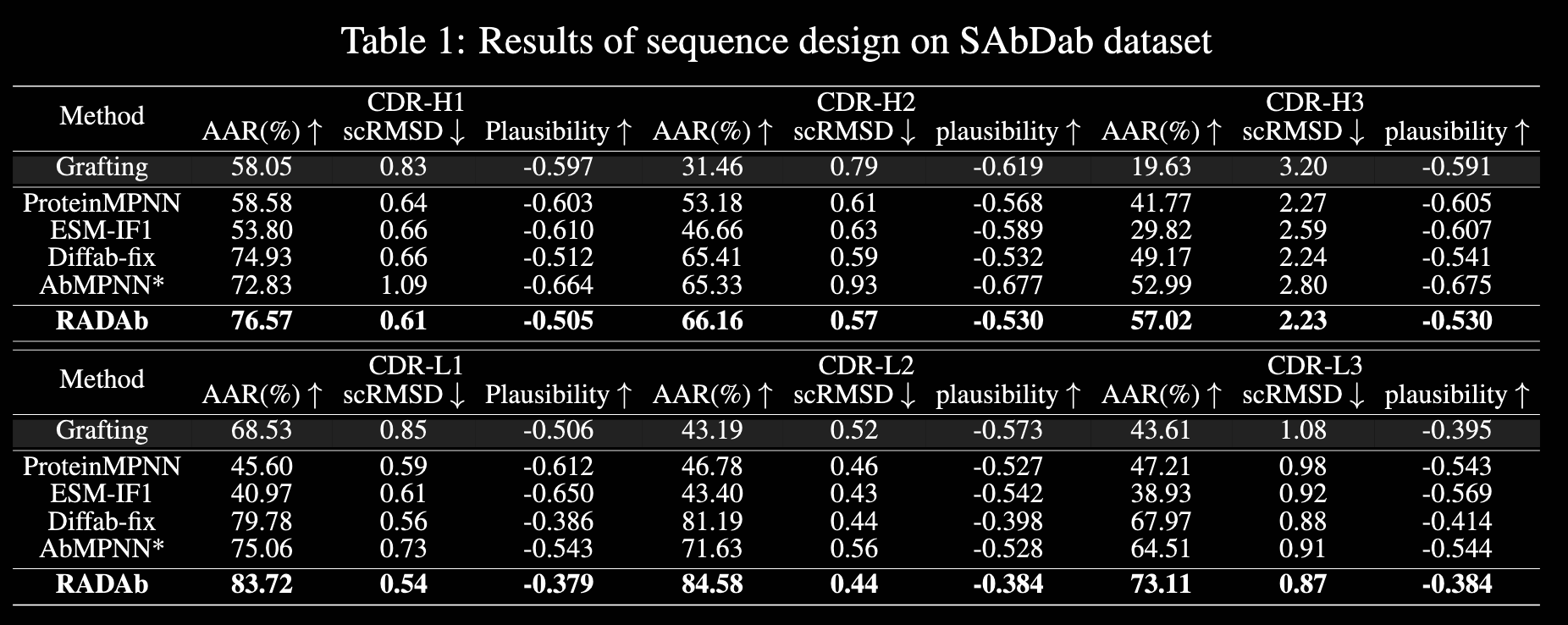
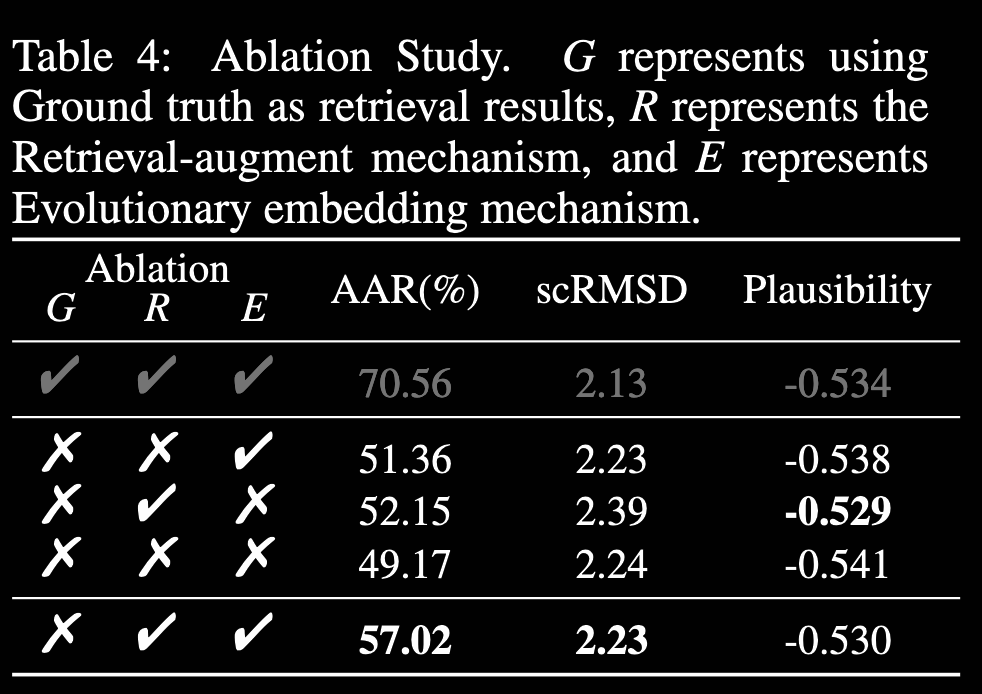
算法由于是用抗原抗体复合物数据,显然也是在SabDab数据上训练的,从结果来看确实相比此前方法有更高的AAR和更低的scRMSD,但是其算法消除实验表明,不出现group truth的搜索结果的情况下,性能会有断崖式下降,这对于不在PDB中存在的全新数据,可能会有明显的影响影响。
同时这篇论文没有对设计结果做必要的湿实验验证,其真实效果有待考察。当然论文作者给我们提供了一个比较新颖的技术路线。抗体数据相对来说较为缺乏,通过PDB中进行信息检索,能够一定程度上弥补这种数据不足带来的影响。